抗体偶联药物(Antibody-Drug Conjugates,ADCs)作为靶向抗肿瘤治疗的重要工具,是一类将单克隆抗体和细胞毒性小分子药物相结合形成的抗体修饰类药物,兼具抗体靶向性和小分子药物杀伤性,该类药物可精准识别杀伤肿瘤细胞,对人体正常细胞的毒性较小[1]。北京协和医学院Zhen等[2]首次从链霉菌C-1027中分离出烯二炔抗肿瘤抗生素力达霉素(LDM),该抗生素由一条酸性辅基蛋白(LDP)和一个九元环烯二炔发色团(AE)以非共价键结合而成,LDP是AE稳定存在的关键所在。研究表明,LDM对肿瘤细胞有着强烈的杀伤作用,且其分子具有可拆分和重建的特点,重建后的分子与天然力达霉素的活性相同[3]。这一特点使得LDM成为构建重组融合蛋白抗体偶联药物的良好材料。
目前,甄永苏院士团队仍致力于开发基于力达霉素的ADCs[4-6],他们通过基因工程和发酵技术制备了Fv-LDP-D3等系列重组融合蛋白,采用分子自组装方法将AE组装到重组融合蛋白上形成ADCs,其中Fv是抗EGFR单链可变区片段,D3是人血清白蛋白(HSA)的结构域Ⅲ。研究结果表明,该类药物对癌细胞具有较好的靶向抗肿瘤活性,但重组后ADCs结构不明,特别是AE分子的组装位置及稳定性尚不清楚。目前,采用冷冻电子显微镜、核磁共振或X射线晶体学等实验技术解析蛋白结构仍有较大挑战性,但随着人工智能技术快速发展,由DeepMind公司开发的 AlphaFold已能够实现对蛋白质进行接近实验精度的结构预测[7]。为此,本文采用蛋白结构预测、分子对接、动力学模拟等方法探讨了该系列ADCs的结构稳定性,期望为基于力达霉素ADCs的优化和开发提供理论依据和数据支撑。
1 计算方法
1.1 蛋白质结构预测
采用AlphaFold3(AF3)[8]对LDP-D3、Fv-LDP、Fv-LDP-D3重组融合蛋白[9]的三级折叠结构进行预测。重组融合蛋白序列如图1所示。通过PyMOL软件对蛋白预测的结果进行可视化[10]分析。
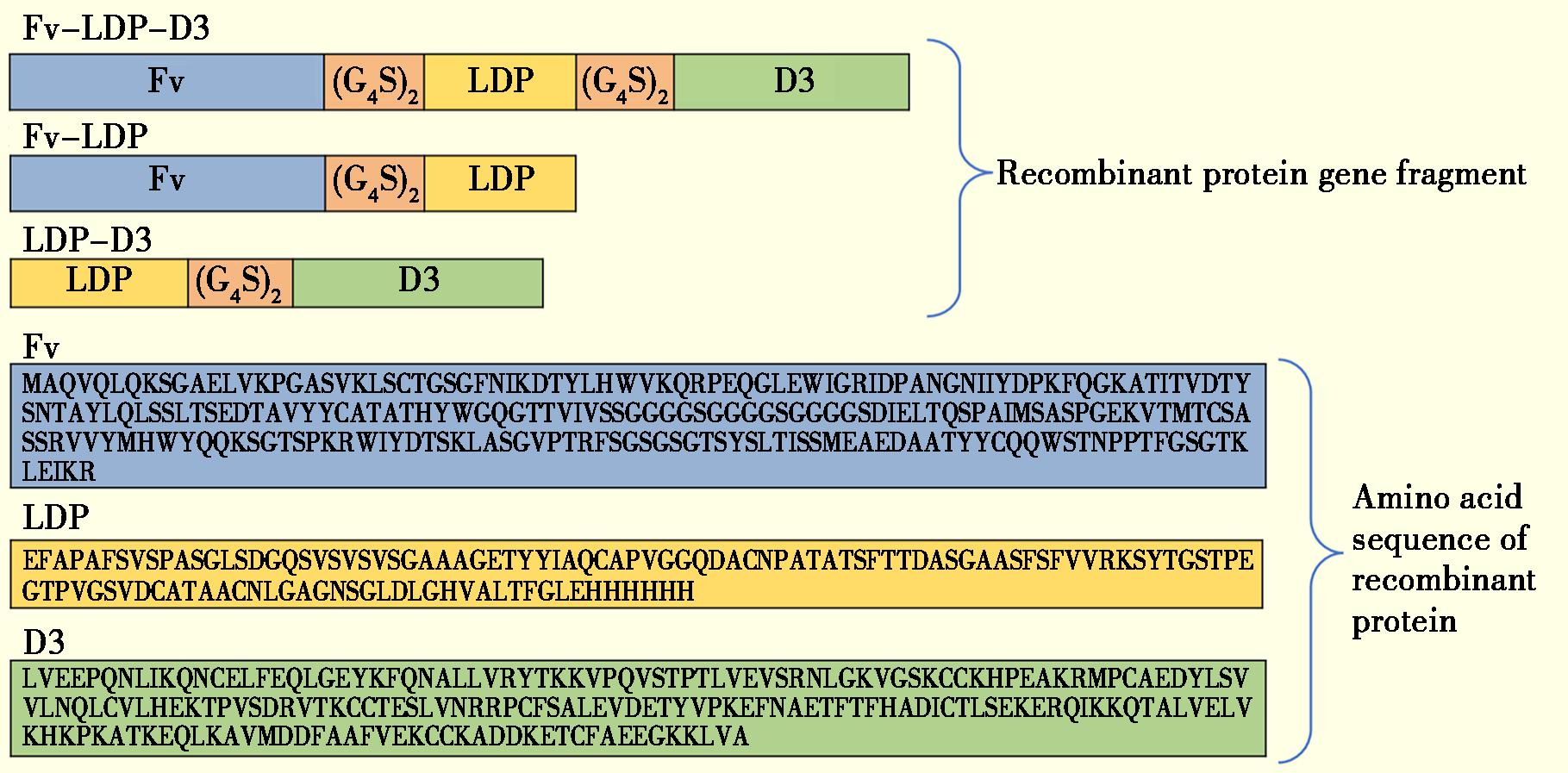
图1 重组融合蛋白序列
Fig.1 Sequence of the recombinant fusion proteins
1.2 分子对接
通过PubChem数据库检索下载AE结构[11],采用ORCA 6.0[12]对其进行结构优化(图2)。采用AutoDock Vina 1.2.0[13,14]程序将配体AE分别与LDP、LDP-D3、Fv-LDP、Fv-LDP-D3对接,并利用基于经验自由能的函数对对接结果进行打分[15],对接盒子设置在含有活性位点的LDP蛋白表面,参数设置见表1,最后用PyMOL软件[16]对结果进行3D可视化处理。
表1 AE与4种蛋白的对接盒子参数设置
Tab.1 Docking box parameters for AE with the four proteins
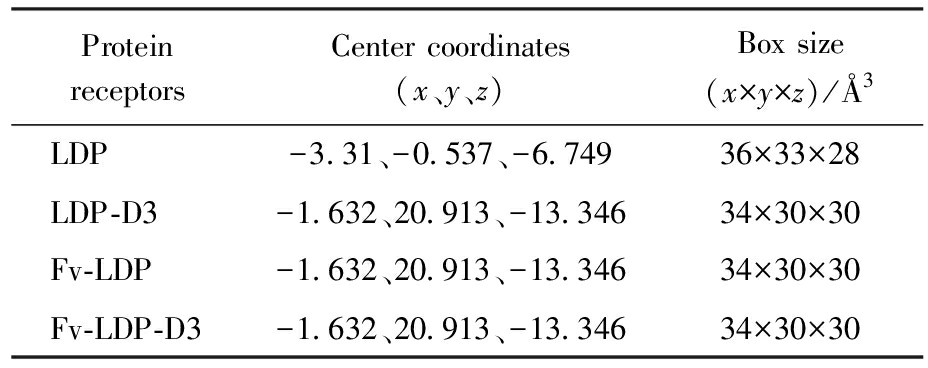
Protein receptorsCenter coordinates(x、y、z)Box size(x×y×z)/Å3LDP-3.31、-0.537、-6.74936×33×28LDP-D3-1.632、20.913、-13.34634×30×30Fv-LDP-1.632、20.913、-13.34634×30×30Fv-LDP-D3-1.632、20.913、-13.34634×30×30
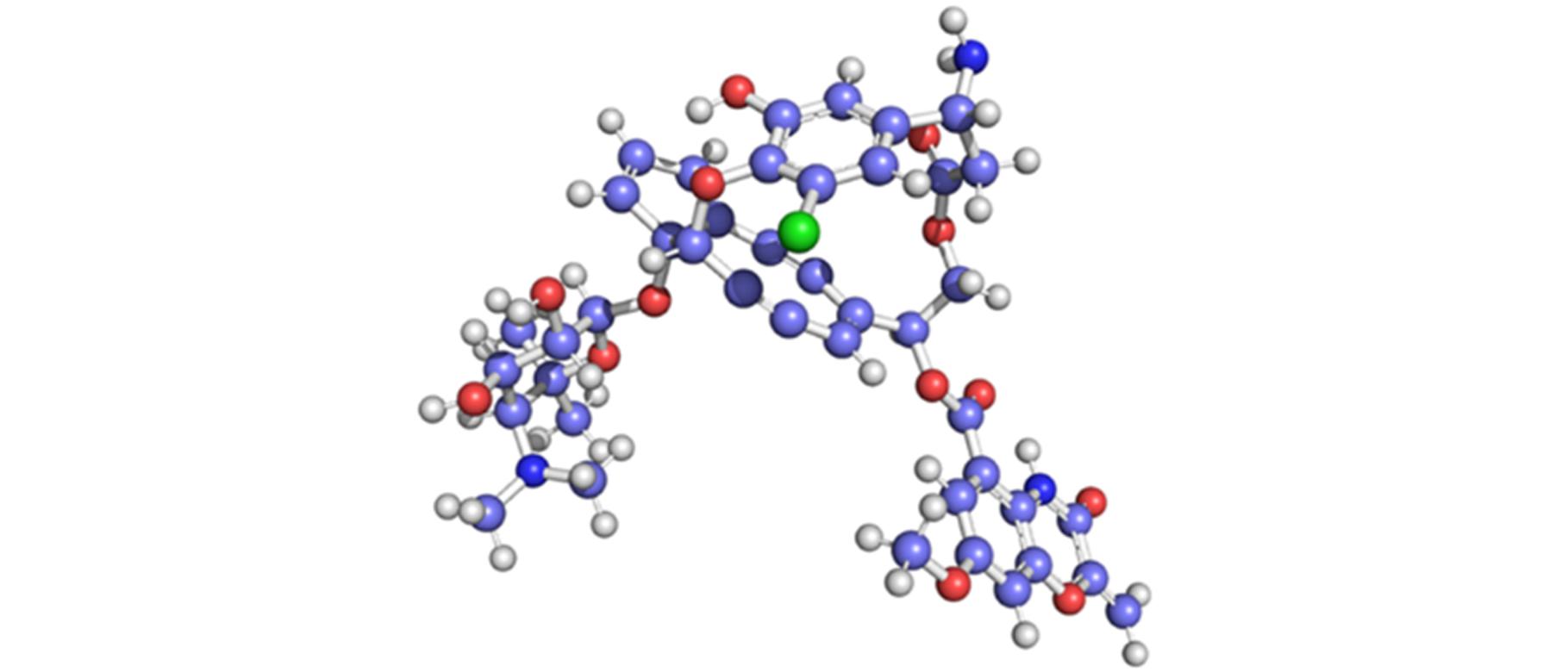
图2 优化后AE的3D结构
Fig.2 Optimized 3D structure of AE
1.3 分子动力学模拟
将分子对接得到的LDP-AE(LA1)、LDP-D3-AE(LA2)、Fv-LDP-AE(LA3)和Fv-LDP-D3-AE(LA4)评分最高构象作为分子动力学模拟的初始结构,模拟采用Gromacs 2024.3[17]软件,蛋白质选择AMBER99SB力场[18],配体采用GAFF力场[19],利用Multiwfn 3.8(dev)[20]、Sobtop 1.0(dev4)[21]生成配体的参数。将体系置于TIP3P显性正十二面体水分子模型中,通过最陡下降法和共轭梯度法将系统能量最小化,NVT系综和NPT系综对其两相平衡100 ps。最后,对整个系统进行100 ns的分子动力学模拟[22]。
2 结果与讨论
2.1 蛋白质结构预测结果分析
选取AF3输出的5个结构中评分最高的蛋白质结构模型[7,8],在PyMOL中进行置信度颜色渲染,结果如图3所示。从图中可见,蛋白结构主体呈红色,平均评分在80~95之间。置信度较低的蓝色部分为无序序列,起连接作用,较为柔性且构象不稳定,可不考虑其影响。该类蛋白质主要由α螺旋和β折叠组成[23],α螺旋主要分布在蛋白质的D3区域,其紧密的螺旋结构使得内部疏水氨基酸侧链相互靠近,形成疏水核心,增强了蛋白质的稳定性,为后续与EGFR的对接提供了保障。β折叠分布在LDP和Fv区域,LDP区域通过折叠形成β片状结构,与小分子AE结合作为ADC药物的重要部位杀伤肿瘤细胞;Fv参与蛋白质之间的相互作用,是靶向EGFR并与其相互作用的重要区域。
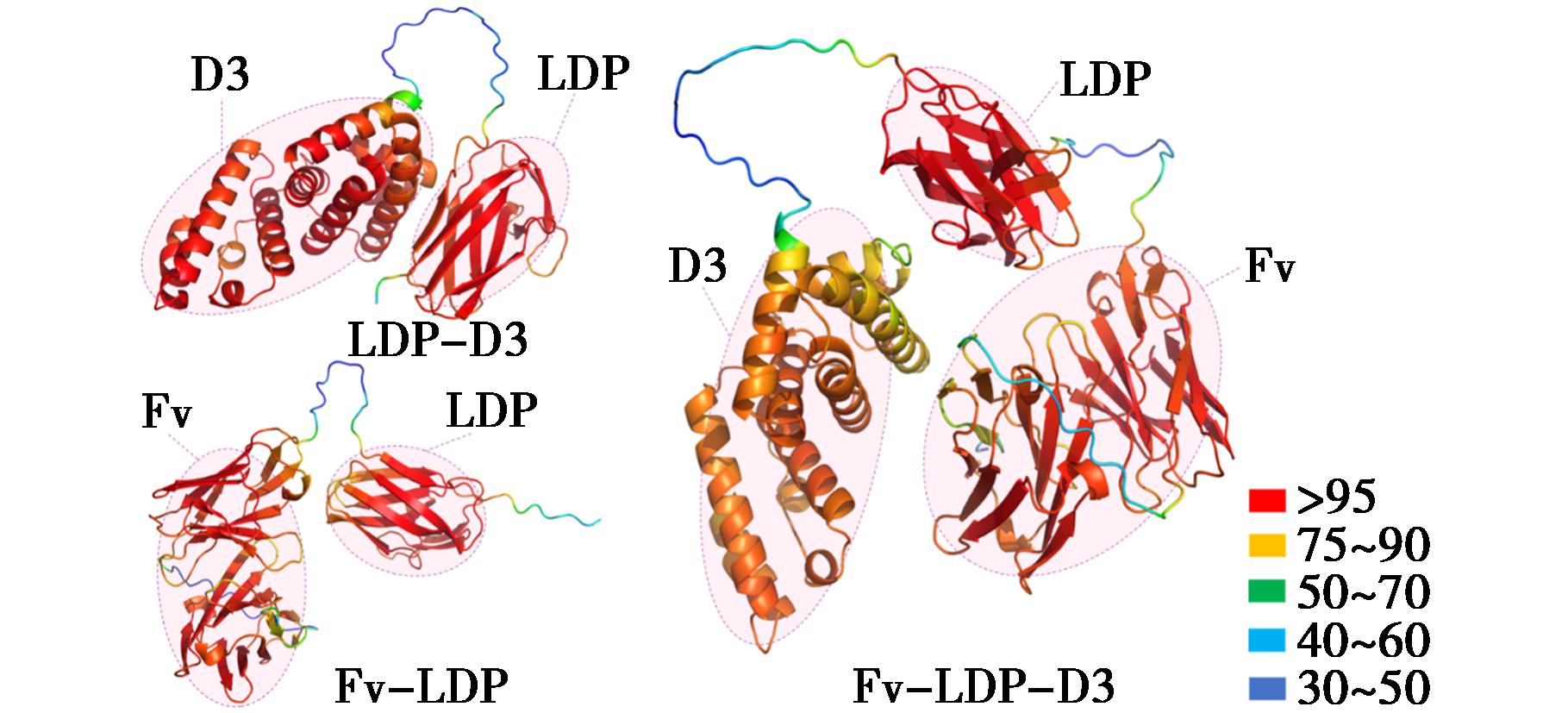
图3 AF3预测蛋白结构置信度渲染图
Fig.3 Confidence score rendering of protein structures predicted by AF3
为了进一步评估AF3对蛋白结构预测的准确性,将预测的蛋白结构Fv-LDP-D3与从PDB数据库中获取的已有蛋白结构进行了分段对比评估(图4)。使用PyMOL计算得出Fv、LDP、D3的均方根偏差(RMSD)。较小的RMSD值说明AF3预测的蛋白结构与PDB库中的实际蛋白结构具有较高的相似性和统一性,为后续分子对接和动力学模拟提供了可信保障。
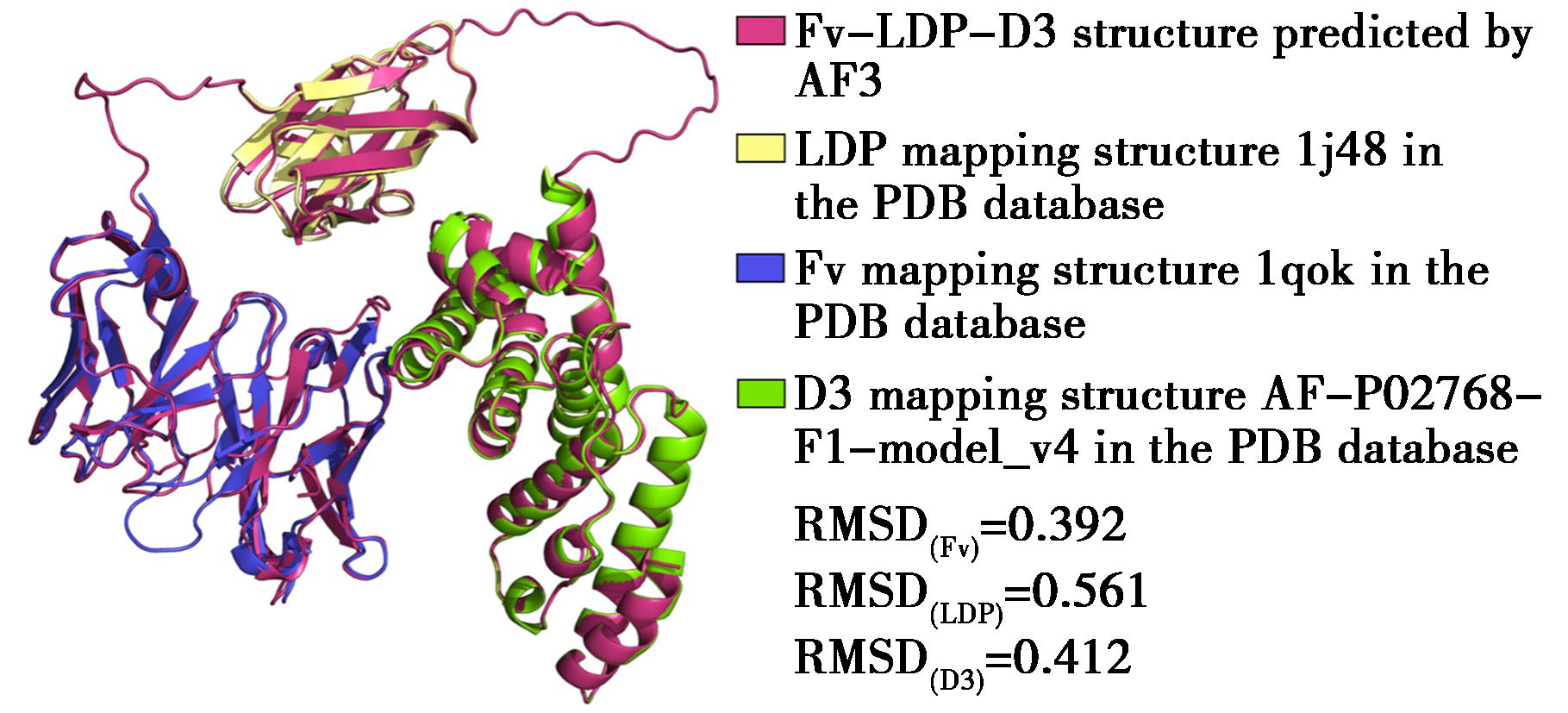
图4 Fv-LDP-D3预测结构与RCSB蛋白质数据库结构的对比
Fig.4 Comparison between the predicted structures of Fv-LDP-D3 and the structures from RCSB Protein Data Bank
2.2 分子对接结果分析
烯二炔发色团AE与4种蛋白LDP、LDP-D3、Fv-LDP、Fv-LDP-D3的可视化结果如图5所示。由图5可知,AE与4种蛋白在LDP表面结合,且相互作用能的主要来源是与LDP表面氨基酸残基形成了氢键和疏水相互作用。表2中列出了对接后与AE配体形成氢键和疏水相互作用的氨基酸残基,以及AutoDock Vina 1.2.0对对接结果的打分。4种复合物的相互作用能均表现为较低的负值,这表明对接结果均具有着较好的稳定性[24]。
表2 AE与4种蛋白的对接结果
Tab.2 Docking results of AE with four proteins
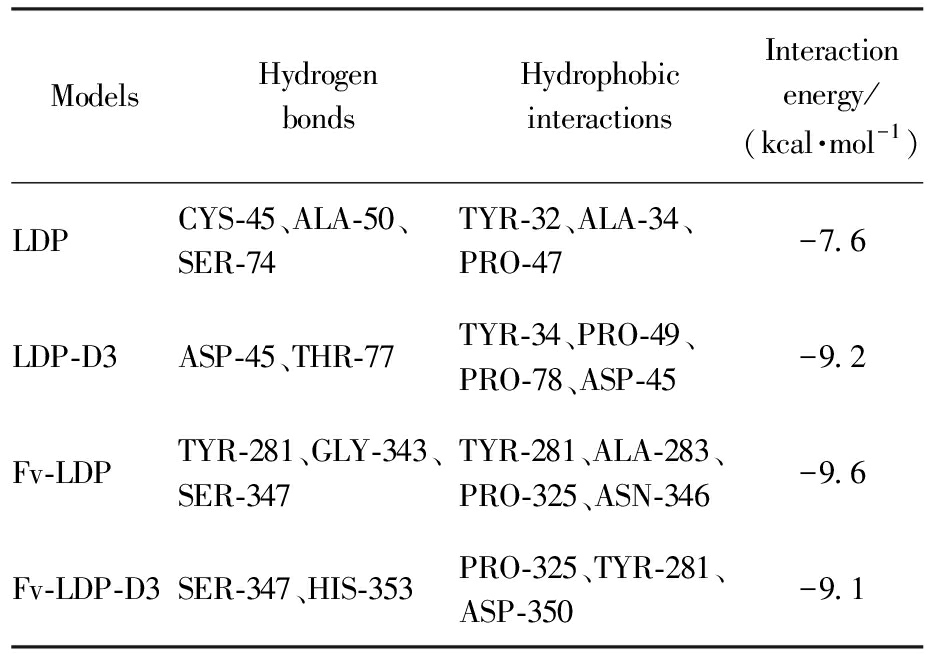
ModelsHydrogen bondsHydrophobic interactionsInteraction energy/(kcal·mol-1)LDPCYS-45、ALA-50、SER-74TYR-32、ALA-34、PRO-47-7.6LDP-D3ASP-45、THR-77TYR-34、PRO-49、PRO-78、ASP-45-9.2Fv-LDPTYR-281、GLY-343、SER-347TYR-281、ALA-283、PRO-325、ASN-346-9.6Fv-LDP-D3SER-347、HIS-353PRO-325、TYR-281、ASP-350-9.1
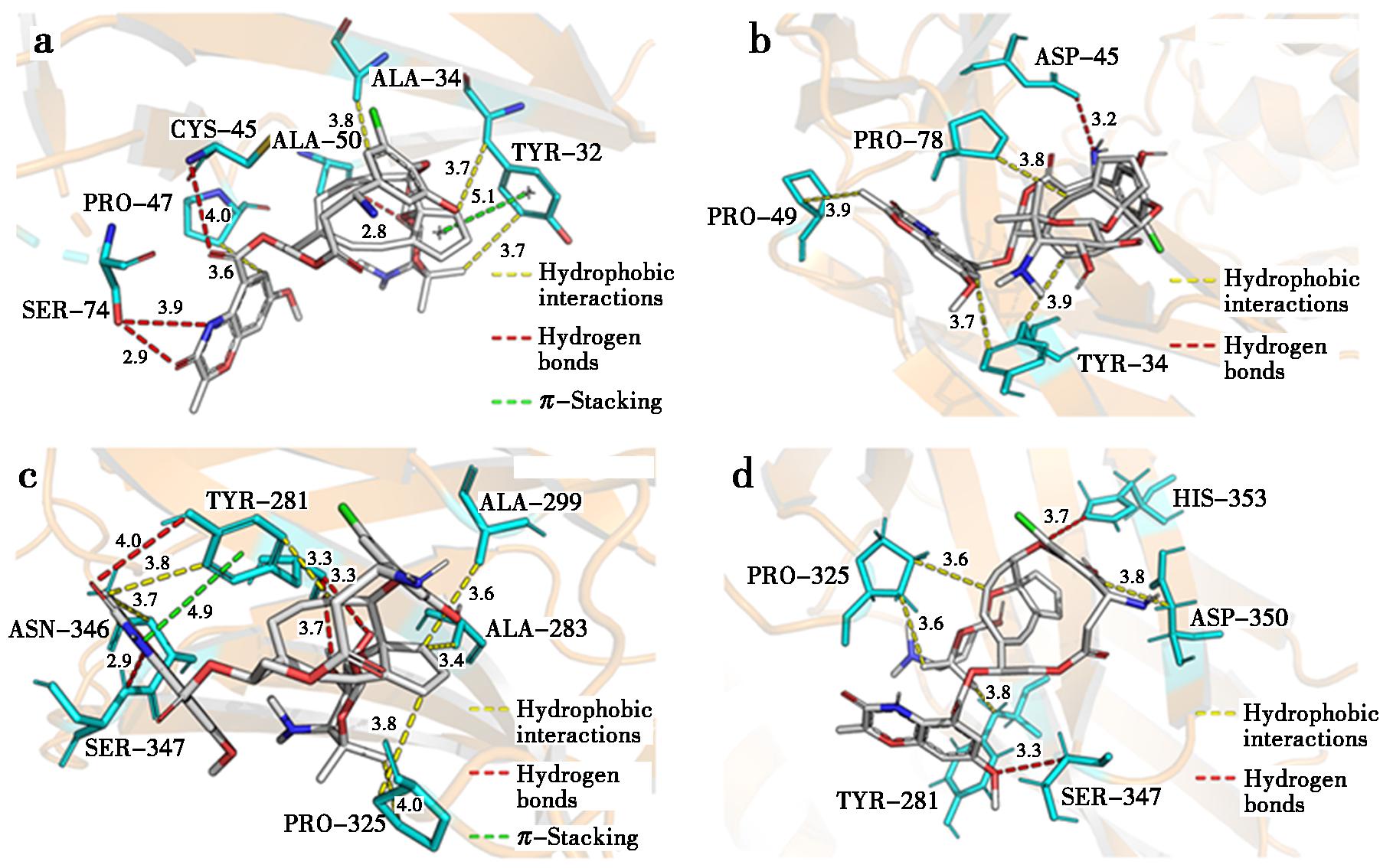
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图5 AE与4种蛋白的可视化对接结果
Fig.5 Visualization of the docking results of AE with the four proteins
2.3 动力学模拟结果分析
2.3.1 均方根偏差分析(RMSD)
RMSD用于评估复合物在模拟过程中相对于初始结构的偏差,反映了体系的稳定性。图6为4种蛋白配体复合物的模拟结构与初始结构之间随时间变化的RMSD。从图中可以看出,力达霉素原始结构达到稳定时间最快,其他重组融合蛋白因蛋白结构增大,稳定时变长,但最终均能达到结构稳定。
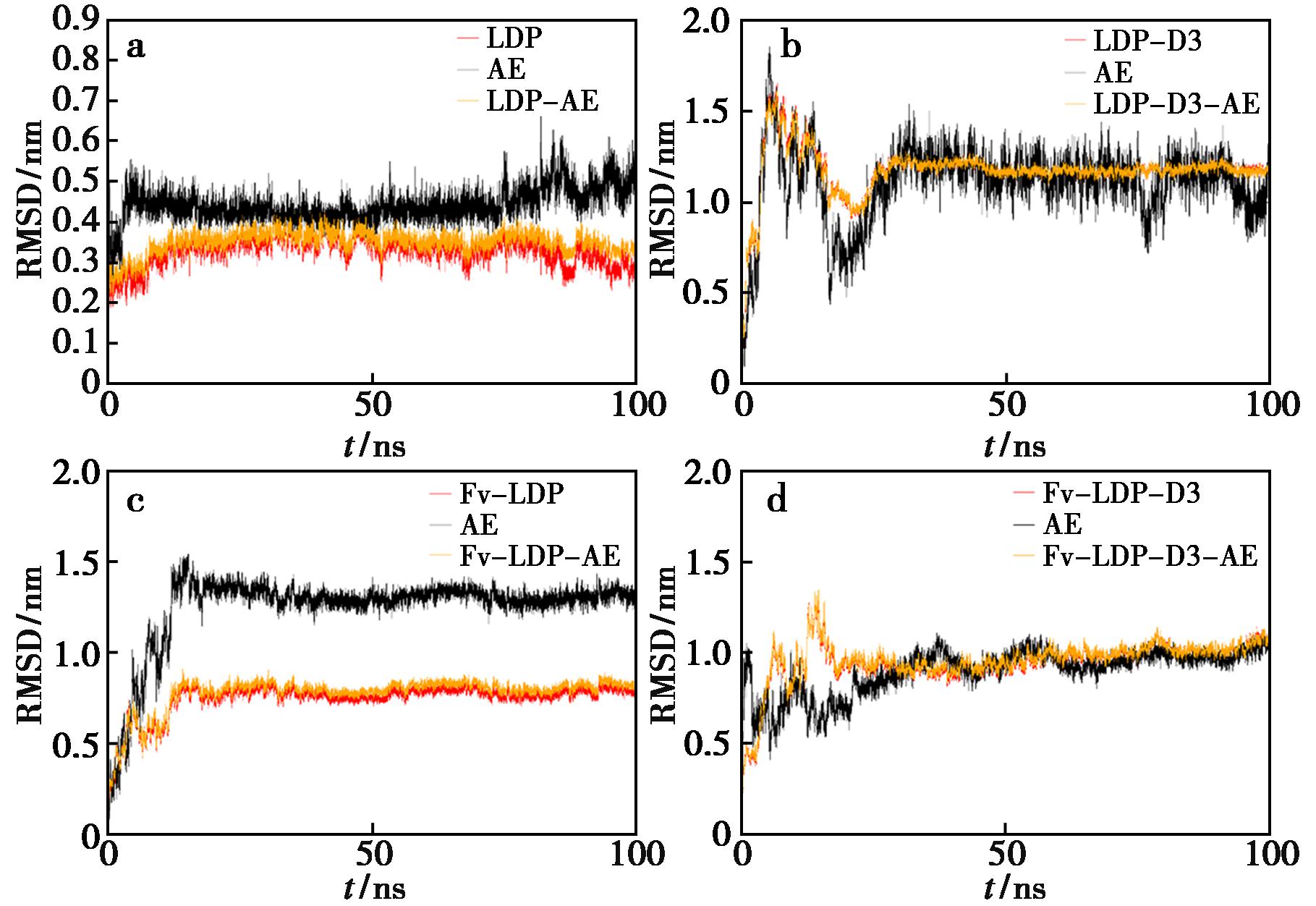
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图6 4种复合物的模拟过程中RMSD的变化
Fig.6 Changes in RMSD during the simulation of the four complexes
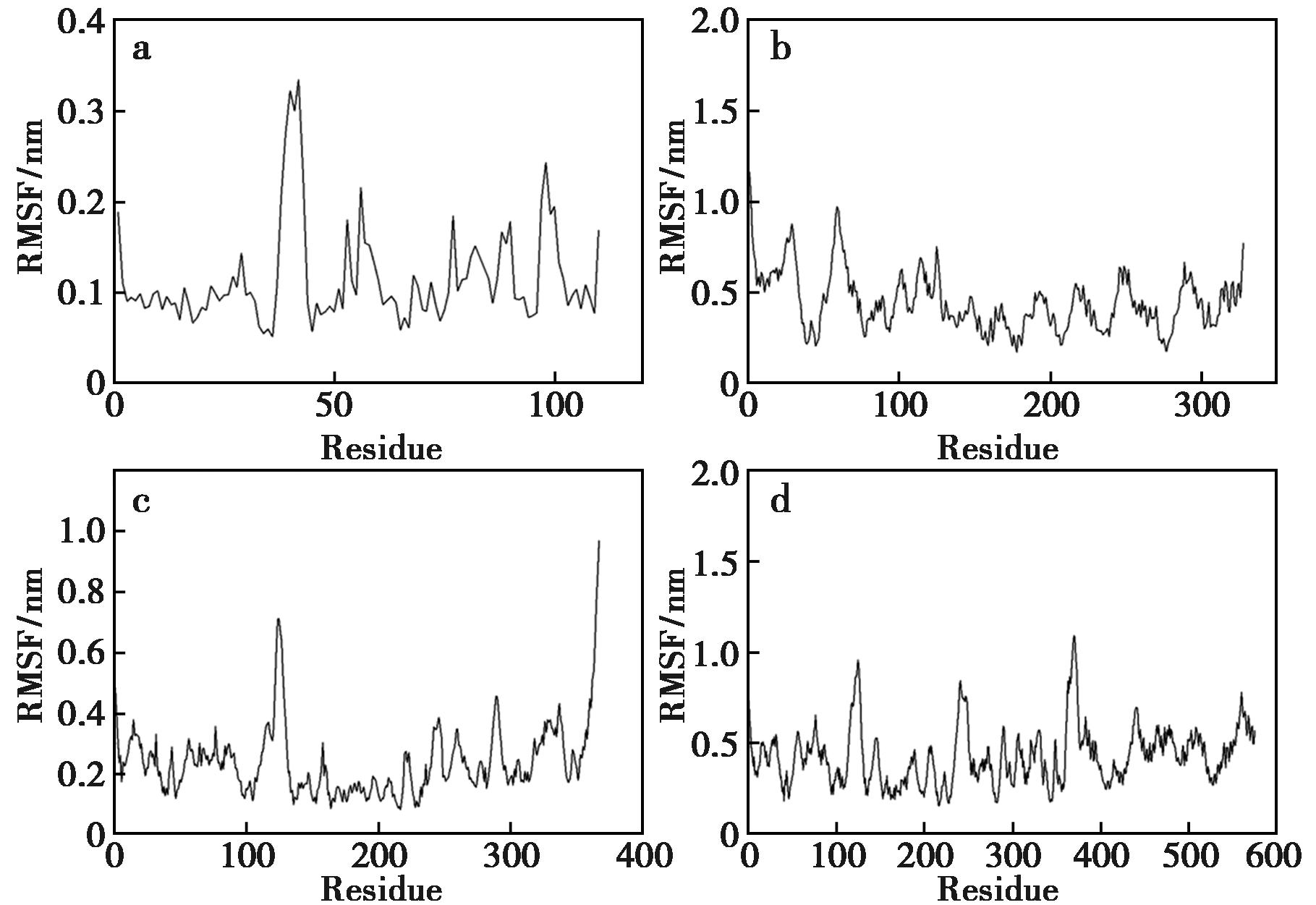
2.3.2 均方根波动分析(RMSF)
RMSF反映氨基酸残基的柔性及波动情况,氨基酸柔性越强,RMSF值越大。图7为4种复合物的RMSF,其中图7a表明LA1的RMSF平均值为0.12 nm,氨基酸残基编号40左右区域波动比较大。分析图7b得LA2的RMSF平均值为0.45 nm,氨基酸残基编号28和58左右区域波动比较大。由图7c可知LA3的RMSF平均值为0.24 nm,波动比较大的数值位于氨基酸残基编号125左右和氨基酸残基末端。图7d表明LA4的RMSF平均值为0.43 nm,波动比较大的数值位于氨基酸残基编号125左右、241左右和370左右区域,表明这3个区域柔性较强。
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图7 4种复合物的氨基酸残基均方根涨落的变化
Fig.7 Changes in root mean square fluctuation of amino acid residues in the four complexes
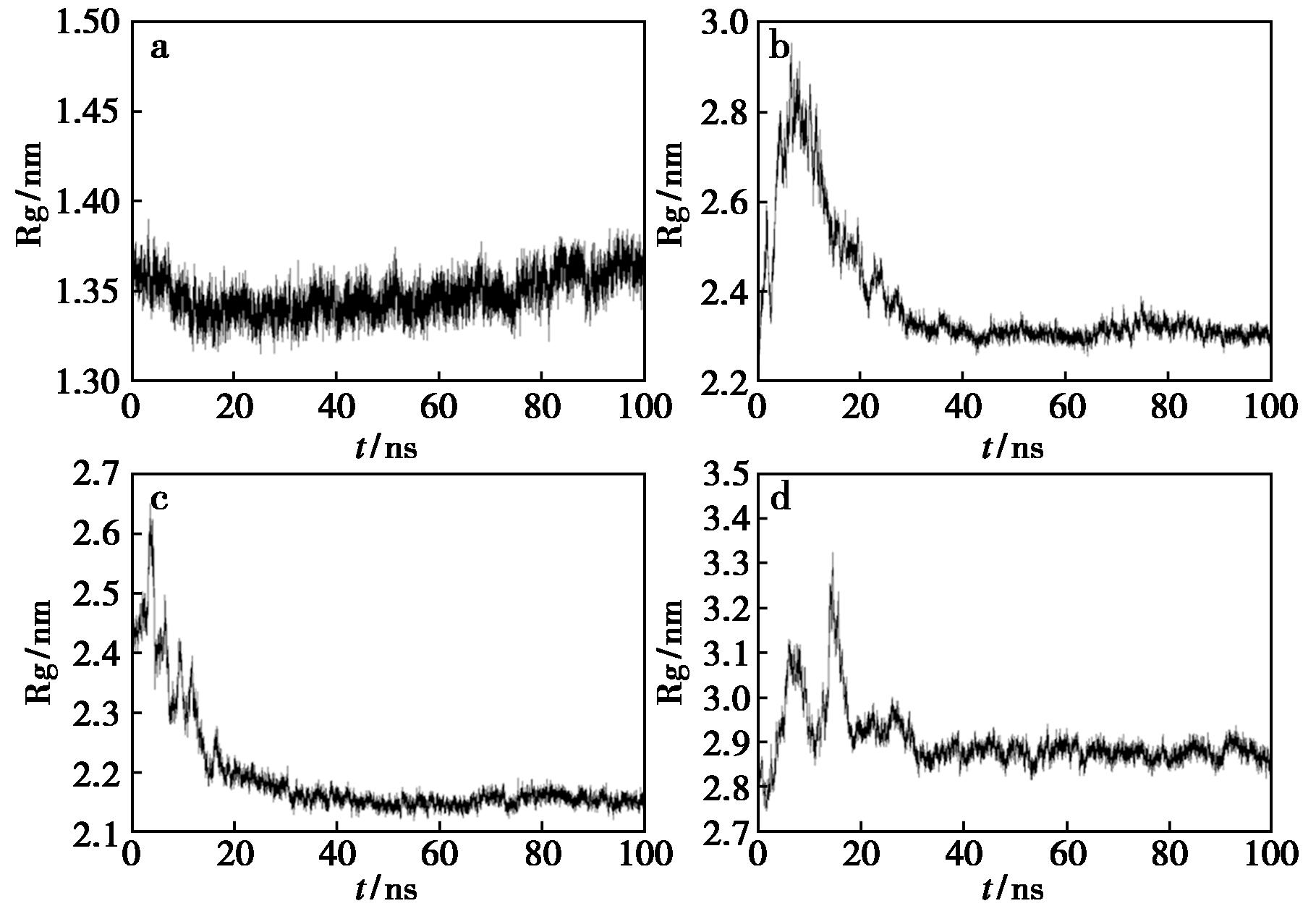
2.3.3 回旋半径分析(Rg)
在动力学模拟过程中,蛋白质的紧凑性、稳定性和折叠程度是由其回旋半径决定的,图8显示4种复合物的Rg随时间的变化。由图8a得LA1的回旋半径波动范围始终稳定在1.347 9 nm左右,表明LDP结构稳定,且与配体紧密结合。分析图8b、8c、8d得LA2、LA3和LA4的回旋半径在 30 ns后趋于稳定,稳定Rg值分别在2.378 9、2.193 1和2.900 9 nm左右,在模拟过程中,蛋白质结构逐渐紧凑。
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图8 4种复合物的回旋半径随时间的变化
Fig.8 Changes in radius of gyration over time for the four complexes
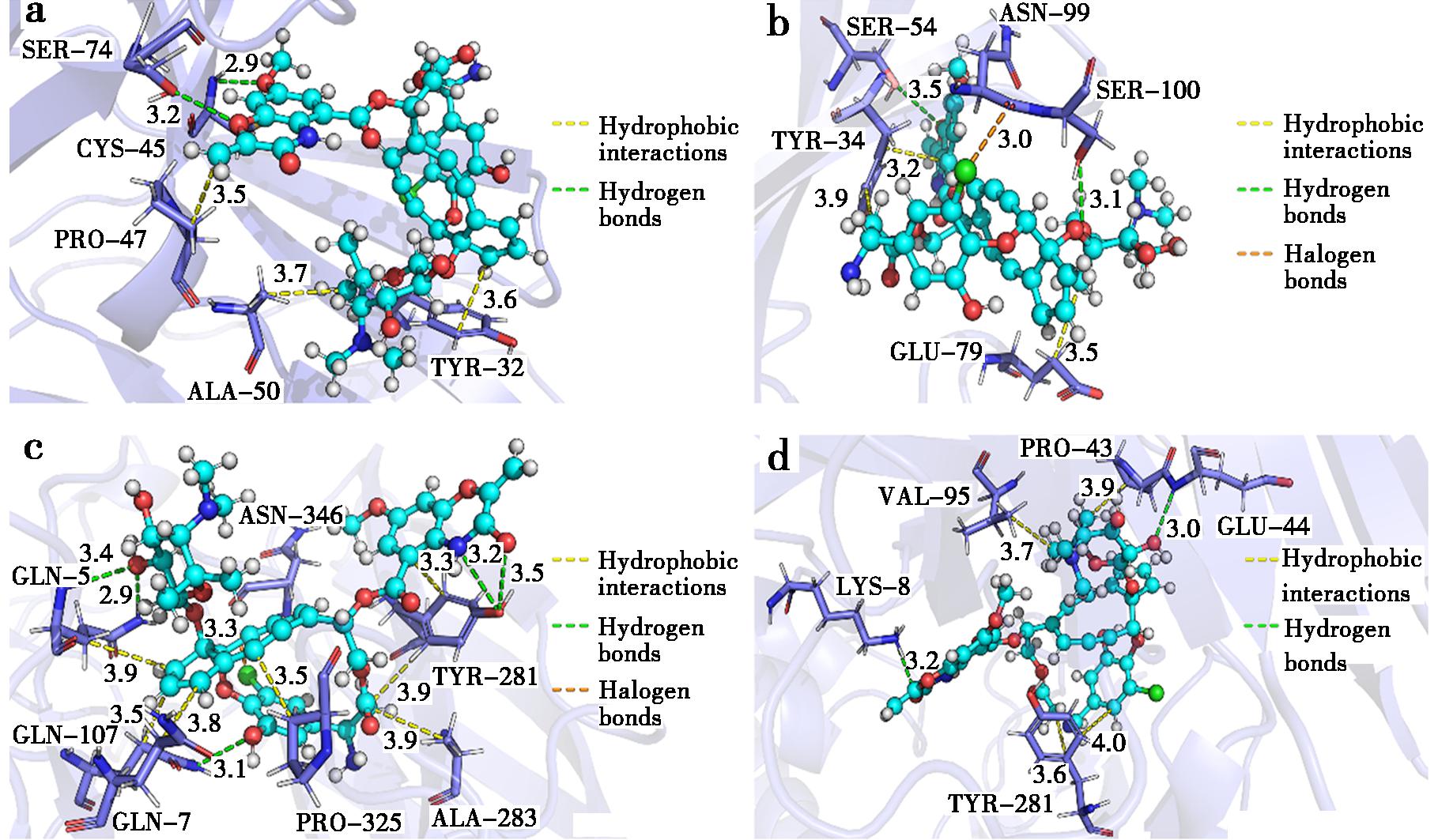
2.3.4 非键相互作用可视化
图9分别为动力学模拟后LDP、LDP-D3、Fv-LDP和Fv-LDP-D3与AE之间的非键相互作用。图9a表示AE与LDP的TYR32、PRO47和ALA50形成疏水相互作用,疏水相互作用作为一种驱动力引导LDP与AE相互靠近,AE与CYS45和SER74形成氢键,使LDP与AE形成稳定复合物。通过图9b可知AE与LDP-D3的TYR34、GLU79形成疏水相互作用,与SER54和SER100形成氢键,并且与ASN99形成卤素键。图9c表示AE与Fv-LDP的CLN5、GLN7、GLN107、TYR281、ALA283和PRO325形成疏水相互作用,与GLN5、GLN107和TYR281形成氢键,并且与ASN346形成卤素键,在Fv-LDP与AE结合过程中,卤素键作为一种吸引力促进二者结合,这些相互作用力使Fv-LDP与AE形成稳定复合物。图5d表示AE与Fv-LDP-D3的PRO43、VAL95和TYR281形成疏水相互作用,与LYS8和GLU44形成氢键,这些相互作用力使Fv-LDP-D3与AE形成稳定复合物。与分子对接结果相比,动力学模拟后稳定构型的疏水相互作用和氢键相互作用氨基酸残基不完全一致。
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图9 4种复合物中蛋白质与AE之间的相互作用力
Fig.9 Interaction forces between the proteins and AE in the four complexes
2.4 结合自由能计算和能量分解
2.4.1 结合自由能计算
采用MM-GBSA[25-27]方法按式(1)~(3)分别计算LA1~LA4的结合自由能。
ΔGbind=ΔEgas+ΔGsol-TΔS
(1)
ΔEgas=ΔEvdw+ΔEele
(2)
ΔGsol=ΔGsurf+ΔGgb
(3)
式中,ΔEgas为气相中的内能,kcal/mol;ΔGsol为溶剂化自由能,kcal/mol;TΔS为熵效应对结合能的贡献,kcal/mol;ΔEvdw为范德华力作用能,kcal/mol;ΔEele为静电作用能,kcal/mol;ΔGsurf为非极性溶剂化能,kcal/mol;ΔGgb为极性溶剂化能,kcal/mol。
本文主要研究蛋白和配体结合的稳定性,因而过程中的熵效应对结合自由能的影响可以忽略不计[28,29]。由表3可知,4种蛋白质与配体AE结合的主要有利贡献是范德华力和静电作用力,相反,极性溶剂化自由能在很大程度上对于4种复合物的结合是不利的。LA1~LA4的结合自由能均为负值,说明对接复合物均会自发形成键合。
表3 通过MM-GBSA计算4个复合物的结合自由能
Tab.3 Binding free energy of the four complexes was calculated using MM-GBSA
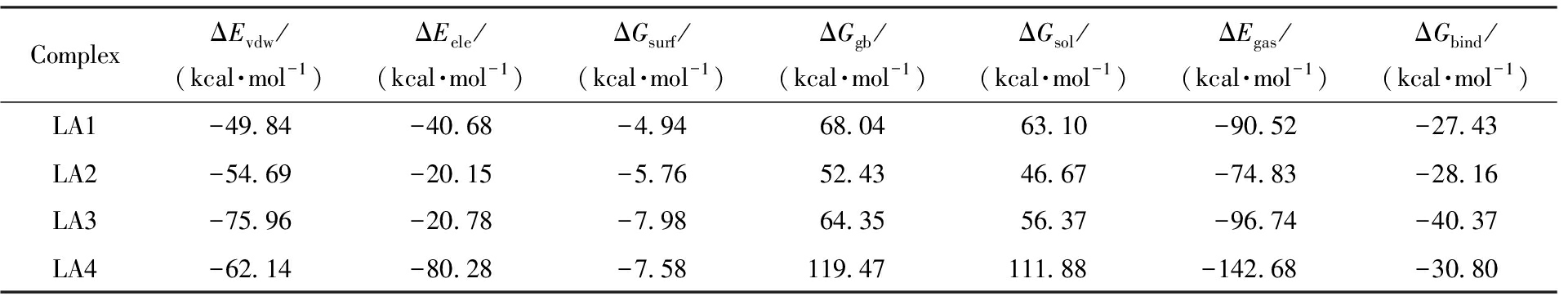
ComplexΔEvdw/(kcal·mol-1)ΔEele/(kcal·mol-1)ΔGsurf/(kcal·mol-1)ΔGgb/(kcal·mol-1)ΔGsol/(kcal·mol-1)ΔEgas/(kcal·mol-1)ΔGbind/(kcal·mol-1)LA1-49.84-40.68-4.9468.0463.10-90.52-27.43LA2-54.69-20.15-5.7652.4346.67-74.83-28.16LA3-75.96-20.78-7.9864.3556.37-96.74-40.37LA4-62.14-80.28-7.58119.47111.88-142.68-30.80
2.4.2 能量分解分析
通过MM-GBSA对氨基酸残基进行能量分解,得到结果如图10所示。图中标注的氨基酸残基能量贡献值均超过1 kcal/mol,这些氨基酸的分布区域与RMSF的分析结果相吻合,说明这些区域是蛋白质的保守区域。分析结果表明在LDP中与AE相互作用的显著残基是PRO76和TYR32,LDP-D3中与AE相互作用的显著残基是TYR34,Fv-LDP中与AE相互作用的显著残基是TYR281,在Fv-LDP-D3中与AE相互作用的显著残基是TYR281和GLN107。
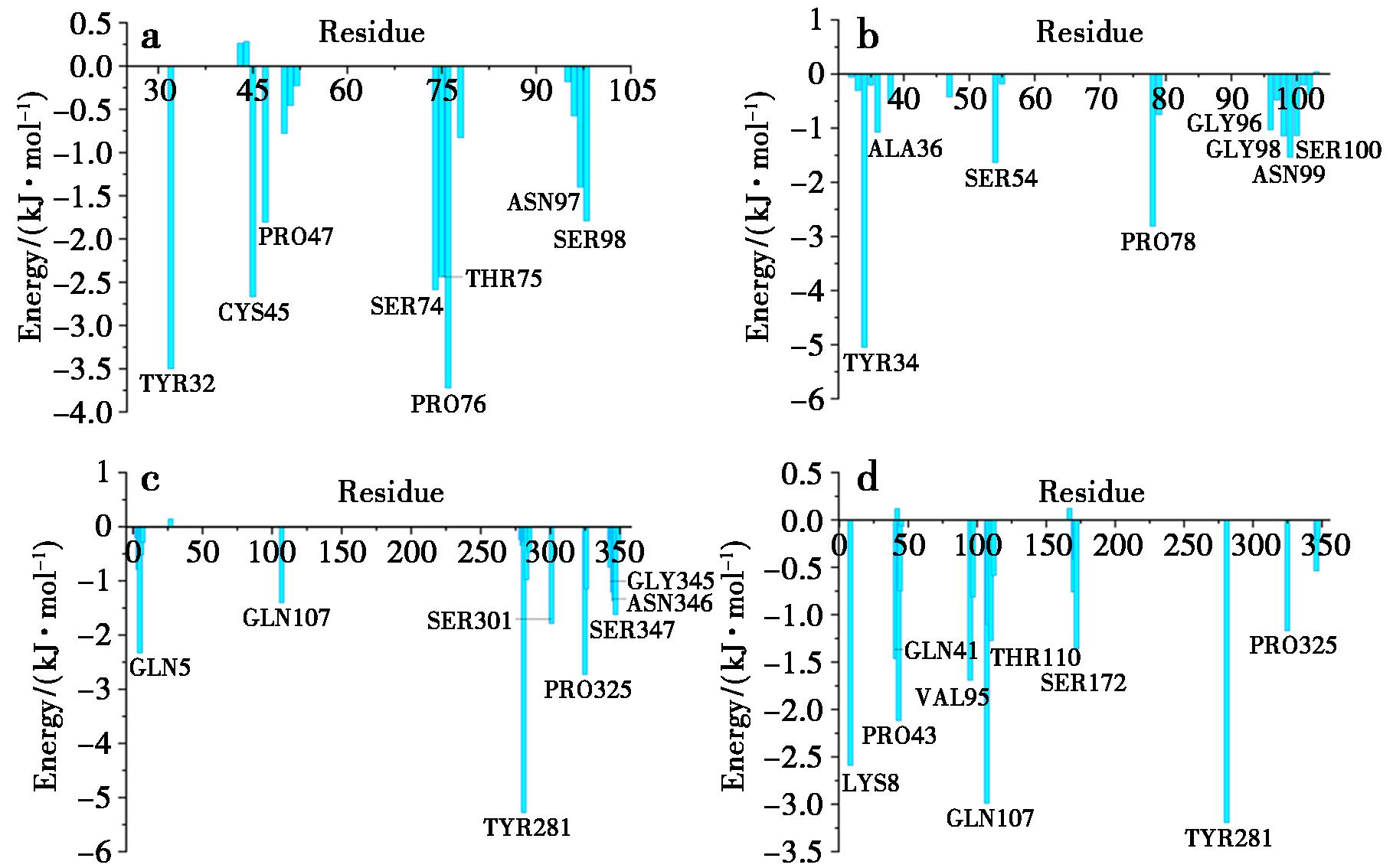
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图10 单个氨基酸残基的能量分解图
Fig.10 Energy decomposition of individual amino acid residues
2.5 独立梯度模型分析
IGM分析是基于电子密度及其梯度信息描述和分析分子间相互作用的分析方法,采用Miltiwfn程序对LA1~LA4进行IGM分析[30,31]。图11所示的红色部分体现了分子间的相互作用,黑色部分体现了分子内的相互作用,四张图中Sign(λ2)ρ显著>0的区域有较多散点分布,说明体系里存在位阻作用,且均在Sign(λ2)ρ约-0.03的位置有1个峰(如箭头所指),由于这些位置电子密度不很大,但又不接近于0,因此可以初步判断是AE与LDP、LDP-D3、Fv-LDP和Fv-LDP-D3之间的氢键[32]。利用VMD1.9.3软件[33]绘制Sign(λ2)ρ填色的δginter等值面图,等值设定为0.003,如图12所示。等值面颜色为蓝色区域的代表氢键,绿色区域代表范德华力。结果说明,范德华力在这4个复合物形成的过程中起着重要作用,氢键作用也有相当一部分的贡献。这与结合自由能计算和非键相互作用可视化中氢键部分的结果吻合。
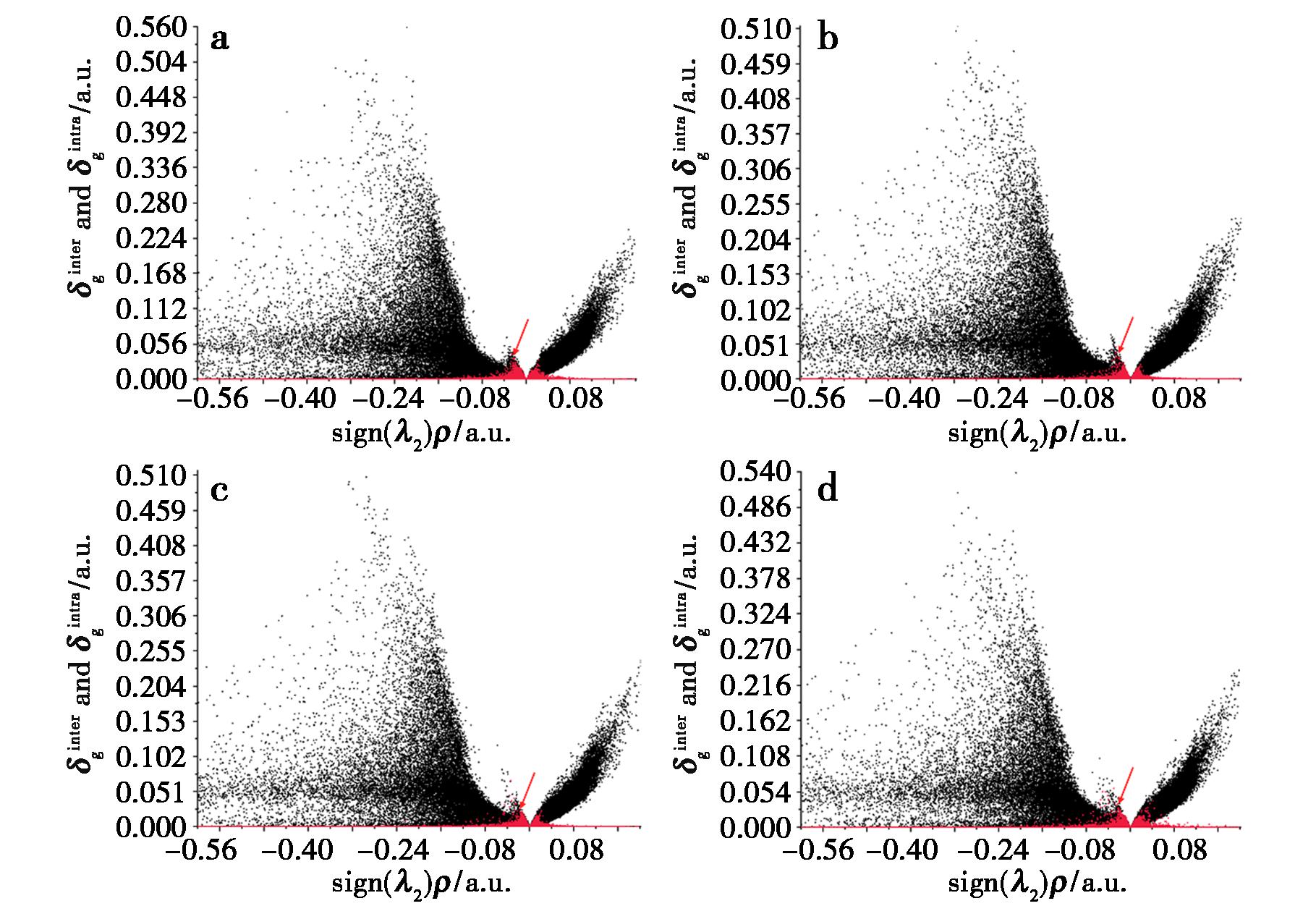
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图11 4种蛋白质与AE的δginter和δgintra对Sign(λ2)ρ的散点图
Fig.11 Scatter plot of δginter and δgintra versus Sign(λ2)ρ for four proteins interacting with AE
a.LDP-AE;b.LDP-D3-AE;c.Fv-LDP-AE;d.Fv-LDP-D3-AE
图12 4种蛋白质与AE相互作用的等值面上色图
Fig.12 Color-mapped isosurface visualization of the interaction patterns between four proteins and AE
3 结论
本文采用alphafold3对Fv-LDP、LDP-D3和Fv-LDP-D3的蛋白结构进行了预测,与已有蛋白结构进行了分段对比评估后,与小分子AE进行了分子对接,对接结果显示4种蛋白质均能与AE形成稳定结构,且均结合在LDP片段部分。以评分最高的对接结果为初始结构,进行动力学模拟,并对模拟结果进行了充分分析,且不同的分析方法之间结果可以相互印证。以上结果均表明,基于力达霉素的重组融合蛋白抗体偶联药物结构在理论上是可以稳定存在的。
[1]Zeng H Y,Ning W J,Luo W X.Chin.J.Biotechnol.,2022,42(5):69-80.曾弘烨,宁文静,罗文新.中国生物工程杂志,2022,42(5):69-80.
[2]Zhen Y S,Ming X Y,Yu B,Otani T,Saito H,Yamada Y.J.Antibiot.,1989,42(8):1 294-1 298.
[3]Shao R G,Zhen Y S.Acta Pharm.Sin.,1995,30(5):336-342.
[4]Wang X,Sheng W,Wang Y,Li L,Li Y,Zhang S,Liu X,Chen S,Zhen Y.Mol.Pharmacol.,2018,15(6):2 403-2 412.
[5]Yu Q,Yuan J L,Zhai X T,Ma J,Miao Q F,Zhen Y S.Acta Pharm.Sin.,2021,56(2):496-502.于群,袁佳璐,翟小田,马健,苗庆芳,甄永苏.药学学报,2021,56(2):496-502.
[6]Sheng W J,Gao R J,Zhen Y S.J.Med.Res.,2014,43(6):25-30.盛唯瑾,高瑞娟,甄永苏.医学研究杂志,2014,43(6):25-30.
[7]Jumper J,Evans R,Pritzel A,Green T,Figurnov M,Ronneberger O,Tunyasuvunakool K,Bates R,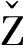 ídek A,Potapenko A,Bridgland A,Meyer C,Kohl S A A,Ballard A J,Cowie A,Romera-paredes B,Nikolov S,Jain R,Adler J,Back T,Petersen S,Reiman D,Clancy E,Zielinski M,Steinegger M,Pacholska M,Berghammer T,Bodenstein S,Silver D,Vinyals O,Senior A W,Kavukcuoglu K,Kohli P,Hassabis D.Nature,2021,596:583-589.
ídek A,Potapenko A,Bridgland A,Meyer C,Kohl S A A,Ballard A J,Cowie A,Romera-paredes B,Nikolov S,Jain R,Adler J,Back T,Petersen S,Reiman D,Clancy E,Zielinski M,Steinegger M,Pacholska M,Berghammer T,Bodenstein S,Silver D,Vinyals O,Senior A W,Kavukcuoglu K,Kohli P,Hassabis D.Nature,2021,596:583-589.
[8]Abramson J,Adler J,Dunger J,Evans R,Green T,Pritzel A,Ronneberger O,Willmore L,Ballard A J,Bambrick J,Bodenstein S W,Evans D A,Hung C C,O′neill M,Reiman D,Tunyasuvunakool K,Wu Z, emgulyt
emgulyt A,Arvaniti E,Beattie C,Bertolli O,Bridgland A,Cherepanov A,Congreve M,Cowen-rivers A I,Cowie A,Figurnov M,Fuchs F B,Gladman H,Jain R,Khan Y A,Low C M R,Perlin K,Potapenko A,Savy P,Singh S,Stecula A,Thillaisundaram A,Tong C,Yakneen S,Zhong E D,Zielinski M,
A,Arvaniti E,Beattie C,Bertolli O,Bridgland A,Cherepanov A,Congreve M,Cowen-rivers A I,Cowie A,Figurnov M,Fuchs F B,Gladman H,Jain R,Khan Y A,Low C M R,Perlin K,Potapenko A,Savy P,Singh S,Stecula A,Thillaisundaram A,Tong C,Yakneen S,Zhong E D,Zielinski M, ídek A,Bapst V,Kohli P,Jaderberg M,Hassabis D,Jumper J M.Nature,2024,630:493-500.
ídek A,Bapst V,Kohli P,Jaderberg M,Hassabis D,Jumper J M.Nature,2024,630:493-500.
[9]He S M.Studies on the Antitumor Efficacy of an EGFR-targeting Recombinant Fusion Protein and its Antibody-drug Conjugate Against Esophageal Cancerand the Mechanism of Action.Beijing:Beijing Union Medical College,2022.何石明.靶向EGFR的重组融合蛋白及其抗体偶联药物的抗食管癌活性和作用机制研究.北京:北京协和医学院,2022.
[10]Chen Y,Zhang H,Wang W,Shen Y,Ping Z.Bioinformatics,2024,40(3):btae139.
[11]Inoue M,Usuki T,Lee N,Hirama M,Tanaka T,Hosoi F,Ohie S,Otani T.J.Am.Chem.Soc.,2006,128(24):7 896-7 903.
[12]Neese F,Wennmohs F,Becker U,Riplinger C.J.Chem.Phys.,2020,152(22):224 108.
[13]Trott O,Olson A.J.Comput.Chem.,2010,31(2):455-461.
[14]Eberhardt J,Santos-martins D,Tillack A F,Forli S.J.Chem.Inf.Model.,2021,61(8):3 891-3 898.
[15]Gaillard T.J.Chem.Inf.Model.,2018,58(8):1 697-1 706.
[16]Delano W L.CCP4 Newsl.Protein Crystallogr.,2002,40(1):82-92.
[17]Abraham M J,Murtola T,Schulz R,P ll S,Smith J C,Hess B,Lindahl E.Software X,2015,1:19-25.
ll S,Smith J C,Hess B,Lindahl E.Software X,2015,1:19-25.
[18]Hornak V,Abel R,Okur A,Strockbine B,Roitberg A,Simmerling C.Proteins:Struct.Funct.Bioinf.,2006,65(3):712-725.
[19]Wang J,Wolf R M,Caldwell J W,Kollman P A,Case D A.J.Comput.Chem.,2004,25(9):1 157-1 174.
[20]Lu T,Chen F W.J.Comput.Chem.,2012,33(5):580-592.
[21]Tian L,Sobtop,Version1.0(dev4) http://sobereva.com/soft/Sobtop/.Accessed on Nov.10,2024.
[22]Su X Y,Huang C,Fan C P,Liu Y Y.Chem.Reagents,2020,42(12):1 403-1 408.苏小燕,黄诚,凡长坡,刘媛媛.化学试剂,2020,42(12):1 403-1 408.
[23]Pauling L,Corey R B,Branson H R.Proc.Nat.Acad.Sci.,1951,37(4):205-211.
[24]Lu J H,Zou Q R,Xiong C W,Qin M,Wu J Y,Li Y X,Cao S S.Chem.Reagents,2024,46(10):1-6.陆俊鸿,邹青容,熊传伟,覃梅,吴甲媛,李映新,曹思思.化学试剂,2024,46(10):1-6.
[25]Valdés-tresanco M S,Valdés-tresanco M E,Valiente P A,Moreno E.J.Chem.Theory Comput.,2021,17(10):6 281-6 291.
[26]Genheden S,Ryde U.Expert Opin.Drug Discovery,2015,10(5):449-461.
[27]Miller B R,Mcgee T D,Swails J M,Homeyer N,Gohlke H,Roitberg A E.J.Chem.Theory Comput.,2012,8(9):3 314-3 321.
[28]Hou T,Wang J,Li Y,Wang W.J.Chem.Inf.Model.,2011,51(1):69-82.
[29]Rastelli G,Rio A D,Degliesposti G,Sgobba M.J.Comput.Chem.,2010,31(4):797-810.
[30]Lefebvre C,Rubez G,Khartabil H,Boisson J C,Contreras-garcía J,Hénon E.Phys.Chem.Chem.Phys.,2017,19(27):17 928-17 936.
[31]Lu T,Chen Q X.J.Comput.Chem.,2022,43(8):539-555.
[32]Lefebvre C,Khartabil H,Boisson J C,Contreras-garcía J,Piquemal J P,Hénon E.Chemphyschem,2018,19(6):724-735.
[33]Humphrey W,Dalke A,Schulten K.J.Mol.Graphics,1996,14(1):33-38.